2020年11月11日,我院任艾明课题组与奥地利因斯布鲁克大学Ronald Micura教授课题组合作在Nucleic Acids Research杂志上发表题为“Structural distinctions between NAD+ riboswitch domains 1 and 2 determine differential folding and ligand binding”的研究论文。
核开关(Riboswitch)是一类保守的非编码RNA分子,位于mRNA 5' UTR区,能够通过识别特定配体如小分子或代谢物而调控下游基因的表达。核开关包含两个功能模块:识别结构域(Sensing Domain)和表达平台(Expression Platform),识别结构域能够特异性性识别并结合特定的配体,从而引起整个核开关包括表达平台构象变化,进而调控下游相关基因的表达。目前已有超过40种核开关被鉴定并报道,配体种类涵盖了金属离子,氨基酸,辅酶以及嘌呤等多种类型的小分子。
烟酰胺腺嘌呤二核苷酸(NAD, Nicotinamide Adenine Dinucleotide),是生物体内的一类重要代谢物,其氧化形式为NAD+,还原形式为NADH。因为良好的电子转移特性,NAD+作为辅酶因子参与细胞内很多重要的氧化还原反应。2019年耶鲁大学的Ronald Breaker教授课题组在nadA基因上游发现了一类保守的能够特异性识别NAD+的RNA结构域,即nadA结构域,也称为NAD+核开关【1】。与其他核开关不同的是,NAD+核开关包含有两个串联的序列相近的独立结构单元Domain1和Domain 2,序列分析表明调控基因表达的表达平台变构序列(SD序列)位于Domain2中,而体外结合实验却发现只有Domain1能够识别NAD+。因而,Domain1与Domain2如何通过与NAD+相互作用调控nadA基因的表达,进而调控NAD+的代谢等这些在新型核开关研究中出现的新问题急需进一步阐明。
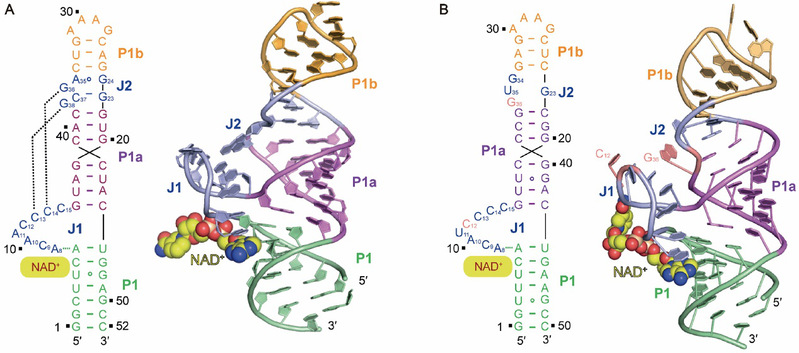
Figure 1. NAD+核开关三维结构
(A) NAD+核开关Domain1结合NAD+的复合物结构; (B) NAD+核开关Domain1结合NAD+的复合物结构。
为了阐明NAD+核开关识别配体的机制以及两个串联Domain对核开关发挥功能的意义,任艾明课题组利用X-射线晶体学的方法,分别解析了 Domain1 (Figure 1A) 和Domain2 (Figure 1B) 结合NAD+的复合物结构。研究结果发现,Domain1和Domain2在三维空间上采用类似的折叠方式,都能够形成NAD+识别口袋,并且在识别口袋中,NAD+分子中的ADP(腺嘌呤二核苷酸)结构部分被RNA分子通过氢键作用、堆积作用以及金属离子的介导特异性识别,而NMN(烟酰胺)结构部分飘在结合口袋之外,与RNA分子主体部分没有直接相互作用 (Figure 2)。
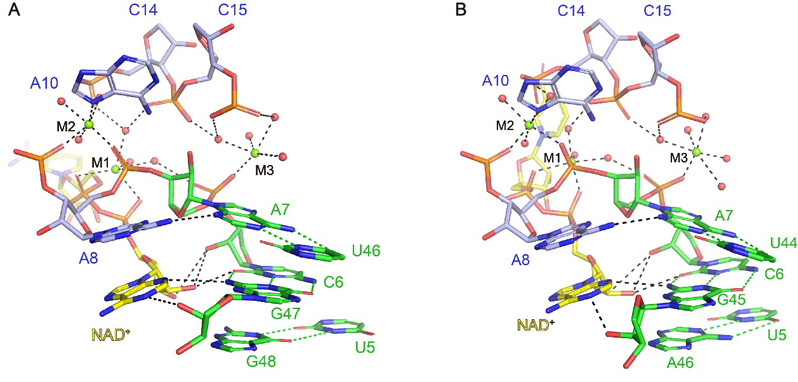
Figure 2. NAD+核开关识别NAD+作用图
(A)NAD+核开关Domain1识别NAD+作用图;(B)NAD+核开关Domain2识别NAD+作用图。
Domain1和Domain2在三维结构及识别NAD+模式上如此相似,又要如何阐释它们与NAD+亲和力之间的差异呢?通过进一步结构比对,我们发现,Domain1的两个环区之间可以形成远程配对作用,而Domain2的两个环区之间无法形成对应的远程相互作用,这可能是导致两个Domain 与NAD+之间亲和力差异的主要因素。随后,我们利用ITC实验等方法证实了我们的猜测。进一步通过荧光动力学分析等方法,我们在实验中观测到Domain1可以在低浓度的NAD+存在下与NAD+结合,而Domain2需要在高浓度(mM级别)的NAD+存在下才能与NAD+结合,进而诱导NAD+核开关表达平台构象的变化,抑制与NAD+合成相关的基因的表达。基于以上实验结果,我们构建了串联结构的NAD+核开关的调控模型 (Figure 3),即Domain1可以低浓度时结合NAD+,传递抑制信号;而当细胞内NAD+浓度达到mM级别时,Domain2对NAD+进行响应,通过与NAD+的结合抑制核糖体与SD序列结合从而抑制翻译。
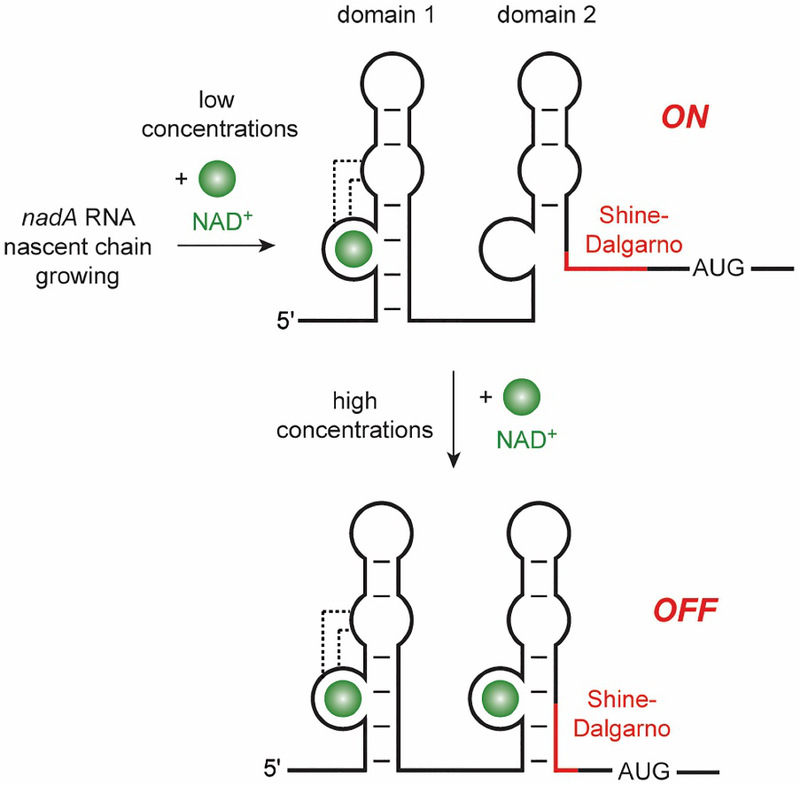
Figure 3. NAD+核开关的调控模型。
此项研究通过解析NAD+核开关的三维空间结构,并提出串联结构的NAD+核开关精密调控基因表达的模型,为进一步探讨复杂RNA分子对重要生命过程的调控机制提供了重要的理论依据和结构基础。
该课题得到了国家自然科学基金(31870810,91940302,91640104,31670826),浙江省杰出青年基金(LR19C050003)以及浙江大学中央高校基金科研项目业务费(2017QN81010)等项目的资助。浙江大学生命科学研究院博士生陈豪和许笑晨为本文的共同第一作者。
参考文献:
1. Malkowski SN, Spencer TCJ, Breaker RR: Evidence that the nadA motif is a bacterial riboswitch for the ubiquitous enzyme cofactor NAD. RNA 2019, 25:1616-1627.
原文链接:https://doi.org/10.1093/nar/gkaa1029