2024年12月16日,我院林世贤课题组合作在ACS Central Science杂志发表了题为“Computationally Assisted Noncanonical Amino Acid Incorporation”的研究论文。该研究通过对常用的氨酰-tRNA 合成酶可识别和难以识别的非天然氨基酸的性质进行归纳总结,开发了一种计算机辅助的非天然氨基酸虚拟筛选器,快速地评估非天然氨基酸被不同种类氨酰-tRNA 合成酶识别的可能性。研究者利用非天然氨基酸虚拟筛选器实现一系列新型赖氨酸和苯丙氨酸衍生物的快速评估和筛选,并成功应用于蛋白质翻译后修饰和调节阳离子-π相互作用。
遗传密码拓展技术利用正交的氨酰-tRNA 合成酶/tRNA 来识别非天然氨基酸,并通过解码终止密码子将其位点特异性的引入到目的蛋白质上。尽管目前已有 300 多种非天然氨基酸被成功应用于各种基础研究和应用研究中,但遗传编码具有独特物理、化学或生物功能的新型非天然氨基酸仍是该领域最具挑战的研究方向。为了实现这一目标,科学家需要设计、化学合成多种具有此类功能的非天然氨基酸,并通过多轮筛选获得具有活性的氨酰-tRNA 合成酶突变体。整个实验流程非常耗时耗力,对于一个专业的实验室需要几个月甚至几年来开发具有特定功能的非天然氨基酸。因此,亟需开发一种高通量的评估策略,在化学合成和氨酰-tRNA 合成酶的筛选流程之前对非天然氨基酸的识别潜力进行评估,并提供非天然氨基酸优化设计方案。
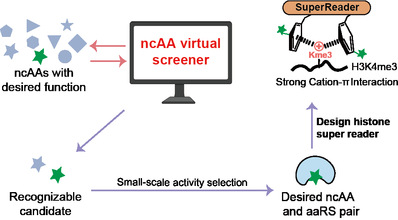
图1 计算机辅助筛选天然氨基酸筛及开发超级阅读蛋白
在这篇论文中,研究者首先对以往报道的 PylRS/tRNA、chPheRS/tRNA、EcLeu/tRNA 和 EcTyr/tRNA 四种正交翻译系统中所有可识别和难以可识别的非天然氨基酸本身的性质以及其与氨酰-tRNA 合成酶的相互作用进行了系统的归纳总结,提出了量化氨酰-tRNA 合成酶对非天然氨基酸识别可能性的参数:1)非天然氨基酸的疏水性:主要影响细胞对非天然氨基酸的摄取效率;2)非天然氨基酸的溶解度:影响非天然氨基酸的胞内浓度;3)非天然氨基酸和氨酰-tRNA 合成酶的亲和力:影响两者的识别。基于这些参数,研究者开发了不同体系的虚拟筛选器。接着,设计了丙二酰化赖氨酸(MalK)和谷氨酰化赖氨酸(GluK)的甲酯衍生物,并通过筛选器验证了其识别潜力,进一步通过实验在大肠杆菌和哺乳动物细胞中实现了遗传编码。最后,研究者们设计了几种具有强富电子的二烷基氨基取代基的苯丙氨酸衍生物,利用chPheRS/tRNA筛选器评估并筛选出了目标非天然氨基酸。这些新设计的非天然氨基酸表现出比酪氨酸和苯丙氨酸更强的阳离子- π 结合能,从而成功开发了组蛋白甲基化超级阅读器。同时,以上研究也证明了,在结合口袋中引入富含电子的Phe、Tyr 或 Trp类似物是设计高亲和力结合蛋白的一种通用策略。
我院博士生方成竹、博士后徐文元和博士后刘超是论文的共同第一作者,林世贤研究员和医学院附属第四医院的丁文龙研究员是本文的通讯作者。该研究受国家重点研发计划,浙江省自然科学基金国家自然科学基金等项目的资助。
原文链接:https://pubs.acs.org/doi/10.1021/acscentsci.4c01544
林世贤实验室聚焦于中心法则的翻译过程,整合交叉学科的研究手段,探索tRNA和蛋白质修饰的生物学功能和工程改造,并致力于开发新型生物医药,用于重大疾病和罕见遗传病的诊疗。欢迎感兴趣的博士后和研究生联系并申请加入,联系邮件sxlin@zju.edu.cn。
实验室链接:http://lsi.zju.edu.cn/25713/list.htm