基因组的三维结构和表观遗传状态在转录调控中起着关键作用,对其深入研究有助于理解细胞命运和疾病发生的分子基础,为精准医学提供有力支持。目前大多数研究都基于群体细胞(bulk cell)的数据,只能反映其平均状态,难以揭示单细胞层面的调控机制。另一方面,尽管单细胞多组学技术已有进展,例如scRNA-seq与scATAC-seq的联合分析方法可同步获取转录活性和染色质可及性信息,但无法捕捉染色质在三维空间中的相互作用;而scRNA-seq与scHi-C的联合分析方法虽可提供染色质互作和基因表达信息,却难以准确解析顺式调控元件与基因启动子之间的特异性互作关系。因此,开发可在单细胞水平同时获取转录、表观遗传和三维结构信息的技术,对于深入理解基因表达的三维表观调控机制至关重要。
为了填补当前的技术空缺,我院阮一骏实验室在前期开发了能在群体细胞水平同时检测染色质相互作用以及染色质开放性的技术ChIATAC[1]。在此基础之上,课题组进一步开发出了具有单细胞多组学分辨率的新技术ChAIR[2]。
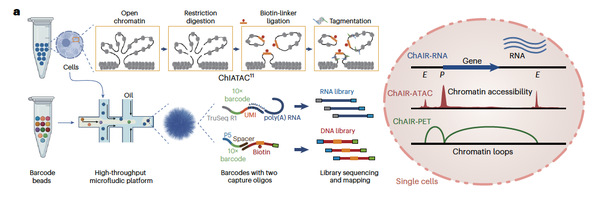
ChAIR技术结合液滴微流控平台,能够在单个细胞中同步捕获染色质可及性(ChAIR-ATAC)、三维结构(ChAIR-PET)和基因表达信息(ChAIR-RNA),单次实验可获取10多万个单细胞数据。其中,ChAIR-RNA可以被用于识别细胞类型、ChAIR-ATAC识别染色质开放性、ChAIR-PET捕获长距离染色质相互作用,一起用于解析细胞类型特异性的三维表观基因组调控网络,系统揭示其与转录激活之间的关系。相比现有三维基因组解析技术,ChAIR在通量和分辨率方面具有显著优势,尤其可在细胞类型特异的水平上精准识别与转录相关的染色质环及高维结构,因而更适用于深入解析三维表观基因组对基因转录调控的机制研究。
借助ChAIR的高分辨率,阮一骏团队分析了35,515个来源于小鼠Patski细胞和222,698个来源于小鼠全脑细胞的单细胞多组学三维基因组数据,发现染色质在有丝分裂和细胞分化过程中,会优先于转录上调建立靶基因与远程顺式调控元件之间的相互作用。这类提前建立的染色质互作常伴随相应位点可及性的增强,最终促使靶基因表达的激活。该时间顺序关系揭示了三维表观基因组与转录激活之间潜在的因果关系。
更进一步,通过将ChAIR-RNA数据与相对应的空间转录组数据相整合,ChAIR还可以揭示空间组织特异的三维表观基因组和转录组特征。相关研究成果已于2025年4月29日发表在Nature Methods,论文题为“Tri-omic single-cell mapping of 3D epigenome and transcriptome in whole mouse brains throughout the lifespan”。
[1] Chai, H. et al. ChIATAC is an efficient strategy for multi-omics mapping of 3D epigenomes from low-cell inputs. Nat. Commun. 14, 213 (2023).
[2] Chai, H., Huang, X., Xiong, G. et al. Tri-omic single-cell mapping of the 3D epigenome and transcriptome in whole mouse brains throughout the lifespan. Nat Methods (2025). https://doi.org/10.1038/s41592-025-02658-7
我院阮一骏实验室副研究员柴皓曦与博士后黄星宇为该论文的共同第一作者。阮一骏教授为论文通讯作者。该研究获得了国家自然科学基金的资助。
原文链接:https://www.nature.com/articles/s41592-025-02658-7