卵母细胞在减数分裂成熟过程中经历长时间的转录沉默,其间的转录组动态变化对卵母细胞质量及早期胚胎发育至关重要,因此受到广泛关注。然而,由于卵母细胞较难获取,因此低起始量需求的Smart-seq2技术广泛被用于检测卵母细胞中的转录本动态变化[1]。然而,在卵母细胞减数分裂成熟过程中,母源转录组的聚腺苷酸化状态发生剧烈变化[2],由于Smart-seq2技术依赖Oligo(dT)引物捕获带有Poly (A)尾的mRNA,这一特性可能导致母源转录组检测产生偏差。
ZAR1和ZAR2是母源转录组稳态调控的关键因子。2019年,Rong等研究人员利用Smart-seq2技术发现ZAR1和ZAR2促进卵母细胞减数分裂成熟过程中母源mRNA的降解[3]。而在2022年,Cheng等研究人员报道ZAR1在GV期卵母细胞中与线粒体协同组装MARDO(Mitochondria-associated ribonucleoprotein domain),并通过Total RNA-seq证实其功能主要为储存和稳定母源mRNA[4]。这两项研究结果在ZAR1对母源转录组的作用方面存在明显差异,因此其在减数分裂过程中的真实调控机制仍有待进一步阐明。
2025年5月9日,我院范衡宇教授团队在Genome Biology杂志上发表了题为“ZAR1 and ZAR2 orchestrate the dynamics of maternal mRNA polyadenylation during mouse oocyte development”的研究论文。该研究联合Smart-seq2、Total RNA-seq、PAIso-seq2[2, 5]和LACE-seq[6]四种近年来开发的、适用于低起始量样本的转录组检测技术,从mRNA稳定性和聚腺苷酸化状态两个维度重新探讨了卵母细胞在减数分裂过程中的转录本动态变化,并深入分析了ZAR1在母源转录组调控中的作用机制。
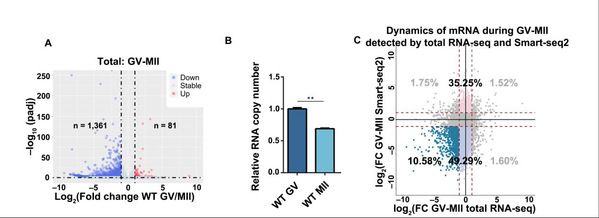
图1:用Total RNA-seq检测母源mRNA动态变化。A:Total RNA-seq检测到的差异基因,MⅡ时期各基因表达水平除以GV时期的表达水平,阈值为Fold change < 0.5或> 2,padj < 0.01。B:ERCC%计算得到的GV和MⅡ时期卵母细胞间的相对RNA拷贝数。C:联合分析Total RNA-seq和Smart-seq2检测的母源转录组动态变化。
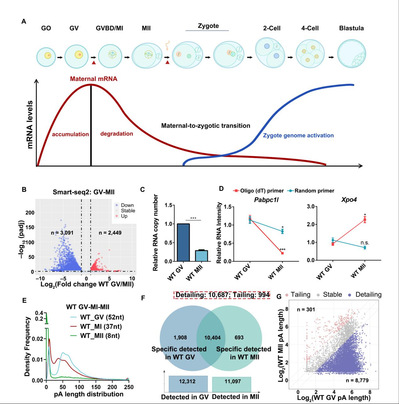
图2:用Smart-seq2和PAIso-seq2检测母源mRNA的动态变化。A:母源转录组动态变化示意图。B:Smart-seq2 检测到的差异基因[3],阈值同图1。C:ERCC%计算得到的GV和MⅡ时期卵母细胞间的相对RNA拷贝数。D:RT-qPCR验证Oligo-d(T)引物和Random引物对GV和MⅡ时期卵母细胞内RNA的逆转录效率。E:PAIso-seq2检测到的各时期卵母细胞中的转录本Poly (A)尾长度分布。F、G:母源转录组差异聚腺苷酸化状态分析。
研究显示,Total RNA-seq检测到的母源转录组在减数分裂过程中的整体下调幅度显著低于Smart-seq2的结果(图1,图2B、C),与此同时,Poly (A)尾长度在减数分裂过程中明显缩短(图2E-G),至MⅡ时期卵母细胞中,平均Poly (A)尾长度仅为8 nt,远低于常用Oligo-d(T)引物的长度。该结果提示,相较于稳定性变化,母源转录组在减数分裂过程中受聚腺苷酸化状态调控的影响更为显著。
此外,该研究重新分析Smart-seq2数据,并结合Total RNA-seq、PAIso-seq2和LACE-seq的结果,进一步明确ZAR1在GV期卵母细胞中通过结合以稳定mRNA,而在减数分裂过程中与其他蛋白协同调控转录本的聚腺苷酸化状态的功能(图3)。
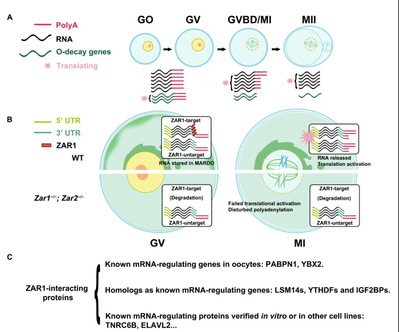
图3:工作模式图。A:聚腺苷酸化状态变化主导母源转录组动态变化。B:ZAR1通过结合mRNAs以调控其稳定性。C:ZAR1与其他蛋白相互作用以调控母源转录组的稳定性和聚腺苷酸化。
综上,该研究通过多组学联合分析,揭示了聚腺苷酸化状态在减数分裂过程中主导母源转录组动态变化的关键作用。研究进一步阐明了ZAR1在卵母细胞减数分裂成熟早期维持母源转录组稳定性,以及在成熟过程中协同其他蛋白调控母源转录本聚腺苷酸化状态的功能。该研究不仅为Smart-seq2技术在卵母细胞中的的适用性提供了参考,也为进一步探索ZAR1蛋白相关的MARDO在减数分裂过程中的作用机制提供了参考资源。
我院范衡宇课题组的博士研究生吴雨珂为本文第一作者。广东省第二人民医院的苏瑞宝副研究员,已毕业博士生蒋知妍和吴韵雯,浙江大学医学院附属邵逸夫医院的戎妍副研究员,浙江大学医学院的嵇姝妍研究员,中国科学院遗传与发育研究所陆发隆研究员及其课题组成员博士生刘静雯和牛卓越,以及中国科学院生物物理研究所薛愿超研究员为该研究提供了大力帮助。该研究受到国家重点研发计划、国家自然科学基金以及浙江省自然科学基金项目的资助。
文章链接见:https://pubmed.ncbi.nlm.nih.gov/40340906/
参考文献