2025年5月28日,浙江大学生命科学研究院汪方炜教授团队在国际著名期刊The EMBO Journal在线发表题为《A chromatin-remodeling-independent role for ATRX in protecting centromeric cohesion》的研究论文,首次发现染色质重塑因子ATRX通过非染色质重塑机制保护着丝粒黏连和染色体稳定性的全新功能,为深入理解癌症等疾病中常见的染色体异常现象提供了关键线索。
有丝分裂是真核生物生长发育的基础。在细胞分裂过程中,姐妹染色单体必须精确分离以确保遗传信息的稳定传递,这一过程依赖于黏连蛋白复合体(Cohesin)介导姐妹染色单体黏连的功能 [1, 2]。黏连蛋白以其环状结构,像“分子胶水”一样将复制后的染色单体紧密“束缚”在一起,直到分裂后期才分开,从而确保染色体的均等分配 [3]。当细胞进入有丝分裂时,染色体臂上的黏连蛋白会被黏连蛋白释放因子Wapl移除,而着丝粒区域的黏连蛋白被保留,以抵抗两极纺锤体的拉力直至分裂后期黏连蛋白被水解酶Separase切断。因此,着丝粒区黏连蛋白的动态调控是染色体正确分离的根本前提。细胞必须进化出严格调控的机制保护着丝粒区的黏连蛋白,以防止在有丝分裂早期纺锤体拉力提前破坏黏连蛋白的功能,导致染色体错误分离,引发癌症等疾病。然而,着丝粒区黏连蛋白的调控机制仍未完全阐明 [4]。
研究团队通过高通量蛋白质组学筛选,首次发现黏连蛋白调节亚基Pds5B直接结合ATRX(α-地中海贫血伴智力低下综合征X连锁蛋白,alpha-thalassemia mental retardation X-linked)。进一步的蛋白互作实验和人工智能预测蛋白结构分析,锁定了ATRX结合Pds5B的关键位点:ATRX的RSYK基序以酪氨酸残基Y1419为核心嵌入Pds5B氨基端的一个疏水口袋。
功能研究发现,突变Y1419残基会显著削弱ATRX与Pds5B的相互作用,导致着丝粒黏连减弱和染色体错误分离,证明了ATRX-Pds5B互作对于染色体稳定性的重要性。超高分辨率显微成像发现,ATRX以异染色质蛋白HP1依赖的方式高度富集于有丝分裂期染色体的着丝粒二分亚结构域(bipartite subdomains),这与近年来所报道的黏连蛋白在此区域的富集相一致 [4]。
分子机制研究发现,ATRX与Wapl通过相似结构基序([R/K]-S/T-Y-x-x)竞争结合Pds5B的相同界面,且ATRX与Pds5B的结合直接阻断Wapl-Pds5B互作,从而抑制Wapl介导的黏连蛋白释放。在细胞内将Wapl敲低后,ATRX对着丝粒黏连的保护作用就变得不再必需。将ATRX的Pds5B结合片段(不含染色质重塑结构域)靶向锚定至着丝粒,可完全恢复着丝粒黏连缺陷,证实其功能独立于ATP酶活性。
论文由此提出着丝粒黏连调控的“双通路”机制(图1):ATRX通路在着丝粒二分亚结构域中拮抗Wapl对黏连蛋白的移除作用,而Haspin通路则在内层着丝粒区(inner centromere)独立调控黏连蛋白的稳定性,两者在保护着丝粒黏连中具有叠加效应。这表明细胞通过多种机制保护着丝粒区的黏连蛋白,从而避免染色体分离错误。
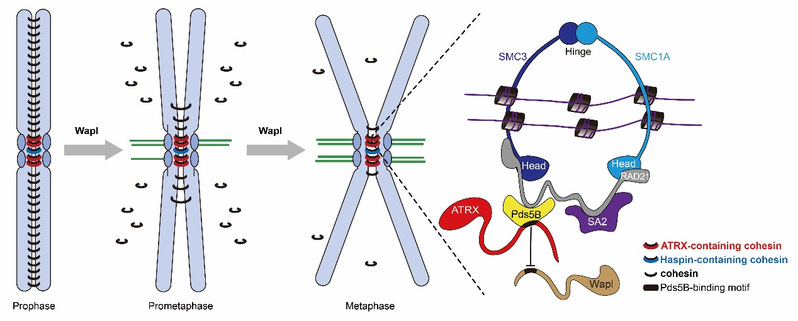
图1:ATRX在拮抗Wapl和维持着丝粒黏连中的作用机制模型。
综上所述,本研究阐明了ATRX通过竞争性抑制Wapl保护着丝粒黏连的功能与分子机制,揭示了其独立于染色质重塑活性的新功能,推动了我们对有丝分裂过程中维持染色体稳定性机制的理解,促进了染色体生物学与癌症生物学研究的交汇。鉴于ATRX在胶质母细胞瘤等肿瘤中的高频突变及其在调控染色体分离中的作用,这项研究对理解肿瘤发生机制及开发靶向黏连蛋白动态的抗肿瘤策略具有重要意义。未来,汪方炜教授团队将探究人类癌症中频发的ATRX基因突变是否会破坏其与黏连蛋白复合体的相互作用,以及这种改变如何导致癌症中的染色体不稳定性,并开发相应的干预策略。
本项目受国家杰出青年科学基金、国家重点研发计划项目、国家自然科学基金和浙江省自然科学基金等资助。我院2021级直博生赵磊为论文的第一作者,汪方炜教授为通讯作者。浙大城市学院颜海燕副教授为共同通讯作者。袁雪颖博士和陈亲富博士提供了重要支持。
原文链接:https://doi.org/10.1038/s44318-025-00465-6
参考文献:
1.Michaelis C, Ciosk R, and Nasmyth K. Cohesins: chromosomal proteins that prevent premature separation of sister chromatids. Cell, 1997. 91(1): 35-45.
2.Guacci V, Koshland D, and Strunnikov A. A direct link between sister chromatid cohesion and chromosome condensation revealed through the analysis of MCD1 in S. cerevisiae. Cell, 1997. 91(1): 47-57.
3.Ochs F, et al. Sister chromatid cohesion is mediated by individual cohesin complexes. Science, 2024. 383(6687): 1122-1130.
4.Sacristan C, et al. Vertebrate centromeres in mitosis are functionally bipartite structures stabilized by cohesin. Cell, 2024. 187(12): 3006-3023.