在真核生物的基因表达调控体系中,染色质结构的动态变化以及多种非典型DNA构象共同构建了高度复杂的转录调控网络。除了经典的右手螺旋B-DNA结构,越来越多的研究表明,多种非B型DNA构象在调控基因表达中发挥关键功能。其中,左手螺旋的Z-DNA是一种通常形成于高转录活性区域的独特DNA结构,被认为与染色质可及性、转录激活以及基因表达稳态等过程密切相关。然而,Z-DNA是如何形成的,其在转录调控中的具体作用机制长期以来仍缺乏系统性的阐明[1,2]。
MYC作为最重要的原癌基因之一,广泛参与细胞生长、代谢与增殖等核心生物过程。MYC能够调节RNA聚合酶II的装载、启动子逃逸及转录延伸,从而影响庞大的基因调控网络。然而,MYC是否能够通过调控DNA构象来重塑转录环境,以及其具体机制为何,尚未得到直接证据的支持。鉴于Z-DNA在高转录区域的富集特征,MYC与Z-DNA之间是否存在功能性关联,一直是相关领域关注的核心科学问题。
近日,我院方东课题组在Nature Communications上在线发表了题为“MYC drives left-handed Z-DNA formation to shape gene expression”的研究论文。该研究首次揭示了MYC驱动Z-DNA形成的分子机制,并进一步阐明了Z-DNA在促进RNA聚合酶II装载过程中的功能作用。研究表明,MYC通过招募染色质重塑因子FACT复合体,使局部DNA由B-DNA发生构象转变形成左手螺旋Z-DNA(图1)。这种构象变化能够显著增强RNA聚合酶II的装载效率,从而精准调控下游基因的转录活性。本研究为理解MYC驱动的转录激活机制提供了全新的结构生物学视角。
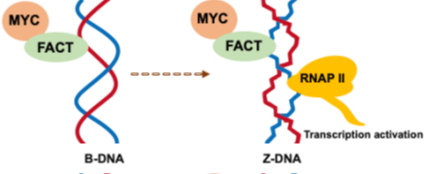
图1:MYC通过FACT诱导Z-DNA的形成模式图
为了系统阐明MYC与Z-DNA的关系,研究团队首先利用免疫荧光实验发现:MYC能够独立于RNA聚合酶II的催化活性促进Z-DNA的形成,并且这一过程依赖于FACT复合体的参与。该发现提示,Z-DNA并非传统意义上由转录活动产生的超螺旋张力“被动塑造”的结构,而是可以通过MYC–FACT被“主动诱导”的染色质构象状态。进一步研究显示,FACT复合体通过重塑核小体中H2A/H2B二聚体,使DNA更易发生构象反转,从而稳定Z-DNA结构的形成。
在功能验证方面,研究团队构建了含人工Z-DNA元件的启动子(Z-SCP)(图2),并通过细胞成像及转录装载实验发现:Z-DNA本身即可增强RNA聚合酶II的结合能力并促进转录启动。该结果直接证明Z-DNA能够作为顺式作用元件激活基因表达,而不仅仅是高转录水平下的伴随现象。
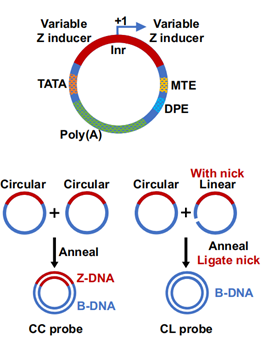
图2:Z-SCP示意图
综上,本研究揭示了MYC通过FACT复合体主动诱导Z-DNA形成并促进RNA聚合酶II装载的分子机制,强调了Z-DNA这一非典型DNA构象在转录调控中的重要功能。该工作不仅深化了对MYC驱动肿瘤发生分子基础的理解,也为开发基于染色质构象的新型肿瘤治疗策略提供了重要理论依据。
方东研究员课题组博士研究生宛心怡、樊浩楠、李卓宁、杜翊瑄为共同第一作者,方东研究员为本文通讯作者。该工作受到上海精准医学研究院黄晶研究员,浙江大学生命科学研究院冯新华教授、黄俊教授、焦会朋研究员、黄德力研究员、曹劲副教授,浙江大学“一带一路”国际医学院宋海教授的大力支持,同时受科技部重大研究计划,国家自然科学基金委,以及浙江大学生命科学研究院的经费支持。
原文链接:https://www.nature.com/articles/s41467-025-66886-3
参考文献
[1] Hubner M.R., Spector D.L. Chromatin dynamics. Annual Review of Biophysics 39, 471–489 (2010).
[2] Bannister A.J., Kouzarides T. Regulation of chromatin by histone modifications. Cell Research 21, 381–395 (2011).
[3] Madhanagopal B.R., et al. The unusual structural properties and potential biological relevance of switchback DNA. Nature Communications 15, 6636 (2024).
[4] Wang A.H., et al. Molecular structure of a left-handed double helical DNA fragment at atomic resolution. Nature 282, 680–686 (1979).