2017年10月30日,生命科学研究院任艾明课题组与奥地利因斯布鲁克大学Ronald Micura教授课题组合作在Nature Communications发表题为"Structure-based Insights into Self-Cleavage by a Four-way Junctional Twister-Sister Ribozyme"的研究论文。任艾明课题组2016级直博生郑路倩同学为本论文的第一作者。
核酶是一类重要的具有催化功能的非编码RNA分子。小的自剪切型核酶(small self-cleaving ribozyme)发现于30多年前,长度在50到150个碱基之间,至今只有九种该类核酶被报道。Twister-sister是于2015年被发现的一种新型核酶。
利用x-射线晶体学的方法,任艾明课题组首次解析了完整的四通道的(four-way junctional)Twister-Sister核酶的三维空间结构。该结构含有五个茎区(Stem)P1、P2、P3、P4和P5, 其中P2、P3、P4和P5由一个四通道的内环(four-stem junction)连接。研究发现,在二级结构中被分散的高度保守性碱基在三级结构中通过长距离相互作用被聚集在整个结构的中心。处于催化位点的两个碱基C62和A63处于茎区P1与P2之间的内环上,并且呈现伸展构象;其中,A63指向内侧并通过堆积作用和氢键相互作用被固定,而C62指向外侧,比较灵活。在催化位点,该项研究还鉴定出了一个非常重要的鸟嘌呤G5,它通过其碱基上的NH1基团直接与催化位点磷酸基团非桥联氧形成氢键,酶切实验发现该碱基突变之后会造成酶活完全丧失。此外,该结构除了鉴定出许多起着结构稳定作用的金属离子,在酶切位点附近也发现了4个镁离子,其中M1通过螯合的水分子与催化位点磷酸基团非桥联氧以及G5均存在相互作用,而其中M2可能会充当广义碱的角色。进一步通过与已报道的三通道(three-way junctional)的Twister-Sister核酶结构以及Twister核酶进行比较,发现该核酶可能了采用一类新的催化机制,该催化机制还有待进一步探索。
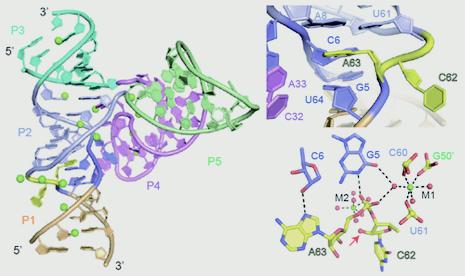
wister sister核酶三维空间结构 Twister sister核酶催化中心构象
此项工作为研究RNA分子的结构特征与核酶的催化机制提供了重要的结构信息,为进一步开发对核酶的应用提供了一定的理论基础。
原文链接:https://www.nature.com/articles/s41467-017-01276-y