2024年6月7日,浙江大学生命科学研究院林世贤实验室在《科学》杂志发表了题为“Rare codon recoding for efficient noncanonical amino acid incorporation in mammalian cells”的研究论文。该研究工作另辟蹊径,提出使用特定的稀有密码子代替空白密码子用于遗传编码非天然氨基酸的新思路,设计并开发了名为“稀有密码子重编码”(Rare Codon Recoding, RCR)的非天然氨基酸编码体系。通过系统的工程改造和核酸序列的大数据模型预测,稀有密码子重编码技术以接近天然氨基酸的编码效率高效合成系列带有非天然氨基酸的功能蛋白质,并在哺乳动物细胞中成功合成带有6个位点非天然氨基酸和4种不同类型非天然氨基酸的蛋白质。
几乎所有生物体的蛋白质都是由20种天然氨基酸组成,这些氨基酸的排列组合形成了种类和功能各异的蛋白质,进而让生物体可以执行复杂的功能。将20种天然氨基酸以外的非天然氨基酸引入到生命体中,将有助于设计并构建出具有全新功能的蛋白质,甚至是全新形式的细胞和生命体。经过几亿年的进化,几乎所有的生命体都在使用同一种遗传密码表,表中的64个密码子已被充分地用于编码20种天然氨基酸,因此理论上没有空间可供非天然氨基酸的编码。
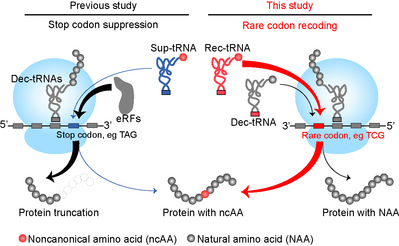
图1. 稀有密码子重编码体系的原理示意图
林世贤实验室成功开发了一种名为稀有密码子重编码(RCR)的非天然氨基酸遗传编码新技术。RCR在原理上与传统的遗传密码拓展技术(Genetic Code Expansion,GCE)不同,通过利用稀有密码子有效地规避了翻译释放因子的激烈竞争,从而解决了哺乳动物细胞中高效编码非天然氨基酸的瓶颈问题(图1)。课题组通过系统的工程改造,建立了新型的大数据模型预测,在哺乳动物细胞中成功实现了以接近天然氨基酸编码效率,高效、高保真度地编码非天然氨基酸。他们还发现RCR技术能够兼容目前常用的几乎所有“正交氨酰-tRNA合成酶/tRNA对”,并利用重编码的重编码翻译系统成功实现了十余种功能非天然氨基酸的重编码。RCR技术的突破理论上可将这300多种非天然氨基酸转移到稀有密码子重编码体系中,显著提升人们设计和合成带有非天然氨基酸蛋白质的能力,并为生产带有特定修饰的大分子产品和药物提供了全新的路线。
浙江大学生命科学研究院博士后丁文龙(已出站)、博士生于微、博士后陈宇霖是论文的共同第一作者,林世贤研究员是本文的通讯作者。研究工作得到了何向伟教授、杨波教授的大力支持与帮助。该研究得到了科技部、基金委、生物医学峰基金等项目的支持。
原文链接:https://www.science.org/doi/10.1126/science.adm8143